近日,中国农业科学院作物科学研究所、四川农业大学小麦研究所/西南作物基因资源发掘与利用国家重点实验室、成都天成未来科技有限公司合作构建了小麦基因定位与基因组研究平台-WheatGmap(https://www.wheatgmap.org),相关研究成果在线发表于《Molecular Plant》(分子植物)杂志上。
据王际睿教授介绍,集群分离分析(BSA)作为一种在分离群体中鉴定目标基因的方法,由于其高效、低成本的优点被广泛应用。然而,对于缺乏生物信息学背景的研究者来说,如何深度分析高通量测序获得的数据、正确选择最优算法、有效利用已公布的海量数据成为当前应用BSA方法进行小麦基因快速定位和候选基因筛选的限速步骤。研发一个界面友好、易操作的专业性数据处理平台将对推动小麦研究有重要应用意义。WheatGmap平台整合了多种基于BSA定位的模型和大量的公共数据,帮助科研工作者利用BSA方法进行小麦基因克隆与功能研究,同时管理与共享测序数据及表型数据(图1)。
图1 WheatGmap页面
WheatGmap目前整合并分析了超过3500份六倍体小麦的高通量测序数据,包括从NCBI、EBI等公共数据库下载的whole-genome sequencing (WGS), whole-exome sequencing (WES), transcriptome sequencing (RNA-seq)数据,以及用户分享的EMS突变体材料测序数据(图2)。为了方便用户利用这些资源,网站同时集成了BSA工具、BLAST功能和基因注释、表达、富集分析。WheatGmap的主要功能模块“Gene Mapping”中整合了四种BSA分析模型,包括SNP-index、Euclidean distance (ED)、QTLseqr和varBScore,研究者以黄绿突变体ygl1分析流程为例介绍了群体构建、数据在线分析、候选筛选等流程。该平台的发布将为小麦研究工作者开展基于BSA的基因定位工作提供方便实用的在线工具。
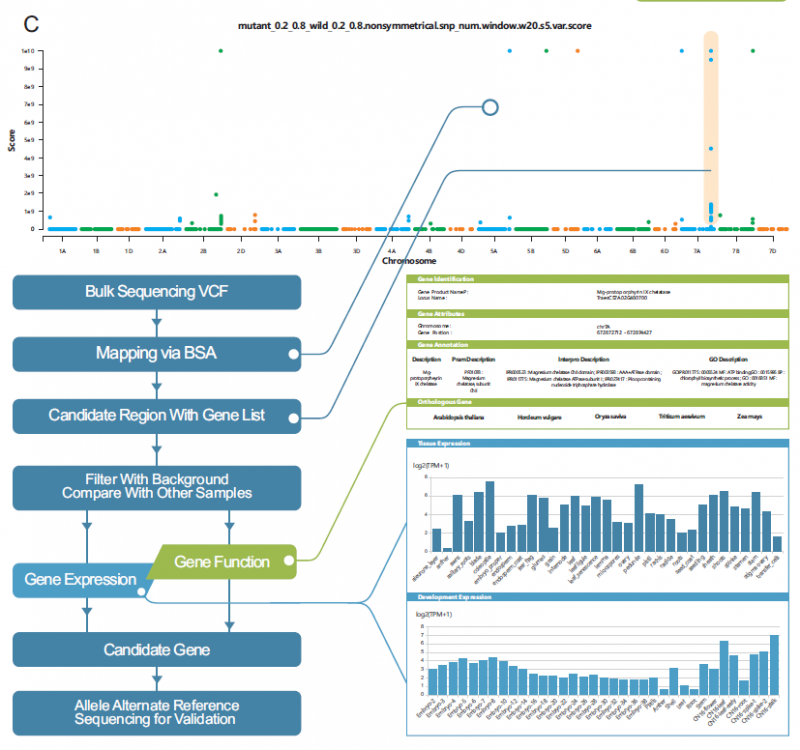
图2基因定位流程、主要结果和关键步骤
中国农科院作科所张立超副研究员、董纯豪博士、四川农业大学博士研究生陈中旭为共同第一作者;四川农业大学王际睿教授、中国农科院作科所刘旭院士、孔秀英研究员为共同通讯作者。该研究得到国家重点研发计划、转基因重大专项、中国农科院科技创新工程、天山创新团队计划和国家自然科学基金、学校双支计划支持。
全文链接:https://doi.org/10.1016/j.molp.2020.11.018



